La Nasa propose depuis 1999 des images MODIS (Moderate-Resolution Imaging Spectroradiometer) à différentes résolutions spatiales et temporelles. Elles sont produites par deux satellites en orbite, Terra (1999) et Aqua (2002), qui embarquent des capteurs pour le programme Earth Observing system.
Les instruments permettent d’enregistrer 36 bandes spectrales avec une fréquence de passage de 1 à 2 jours. La résolution des images diffère selon les bandes enregistrées et varie entre 0.25 et 1 km. Nous parlions il y a quelques jours du site reverb|echo de la Nasa, il se trouve qu’un certain nombre d’images sont disponibles sur le site.
Quelles données ?
Mais pour ça il faut savoir ce qu’on cherche, et la nomenclature des produits n’est pas vraiment transparente [1]. À titre d’exemple :
- MOD9Q1 – Surface Reflectance 8-Day L3 Global 250m
- MOD13Q1 – Vegetation Indices 16-Day L3 Global 250m
- MOD11A2 – Land Surface Temperature/Emissivity 8-Day L3 Global 1km
Une fois qu’on en sait un peu plus sur le type de produit que l’on veut utiliser, il va falloir le télécharger et le traiter. Dans un post précédent, j’expliquais comment configurer sa machine pour utiliser wget. Si vous optez pour cette solution, il vous faudra utiliser le MRT pour reprojeter et assembler vos images. L’interface est assez intuitive et on peut faire du traitement de masse.
Mais je suis tombé aussi ce matin sur les logs de release d’un package R que je me suis empressé de tester : MODIStsp. Si vous avez déjà regardé MRT, MODISStsp n’est qu’un moyen de rester dans R pour télécharger les images. On pourra aussi regarder du côté du package MODIS qui lui n’intègre pas d’interface graphique.
Bon mais on parlait de NDVI dans le titre !
Traiter les images NDVI : MOD13Q1
En cherchant comment traiter les images MODIS, je suis tombé sur des ressources intéressantes. Notamment une page qui vous prend par la main pour traiter les données MODIS (MOD13Q1) dans R[2]. Je vous propose donc de faire un petit point pas à pas !
J’ai téléchargé et projeté les données grâce au package MODISStsp. J’ai donc un dossier qui contient les fichiers hrf, mais aussi un autre qui contient les fichiers tiff produits par le package.
Or quand on ouvre les données (dans Qgis), les gammes de valeurs ne correspondent pas à celles attendues pour du NDVI! On devrait avoir des valeurs entre -1 et 1 !
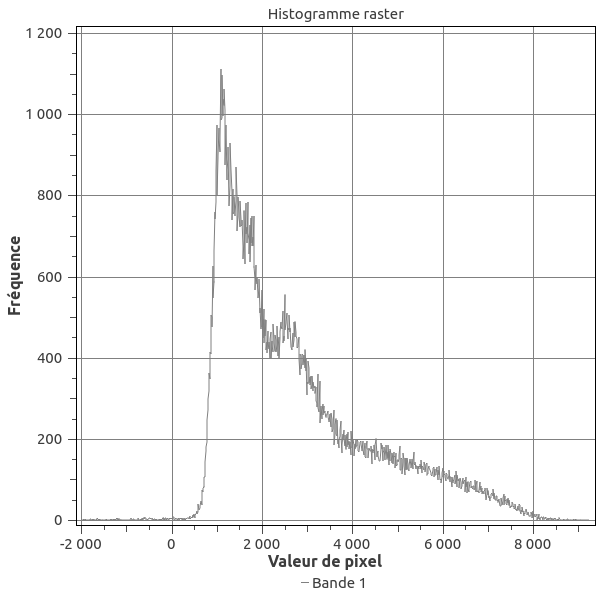
Il va falloir retravailler le raster pour obtenir les bonnes valeurs . Heureusement « the office of outer space affaire » nous donne la marche à suivre dans R[3]. Je vous propose donc ici un script commenté et customisé pour l’occasion.
library(raster)
library(sp)
library(doParallel)
library(rgdal)
library(magrittr)
rm(list = ls()) ##clear env.
#create stack from tif NDVI files
path.v = "~/Documents/futurSahel/MOD13Q1_TS/VI_16Days_250m_v6/NDVI/"
l.files = list.files(path.v, pattern = ".tif")
my.stack = stack(paste0(path.v,l.files)) ##create raster stack of files in a directory
# load borders
border = shapefile("~/Documents/futurSahel/Senegal_gadm.org/SEN_adm0.shp") #ToDo: insert link to the shapefile with the country borders
## We use doParallel and magrittr packages to pipe different actions (crop and mask)
registerDoParallel(6) #we will use 6 parrallel thread
result = foreach(i = 1:dim(my.stack)[3],.packages='raster',.inorder=T) %dopar% {
my.stack[[i]] %>%
crop(border) %>%
mask(border)
}
endCluster()
ndvi.stack = stack(result)
ndvi.stack = ndvi.stack*0.0001 #rescaling of MODIS data
ndvi.stack[ndvi.stack ==-0.3]=NA #Fill value(-0,3) in NA
ndvi.stack[ndvi.stack<(-0.2)]=NA # as valid range is -0.2 -1 , all values smaller than -0,2 are masked out
names(ndvi.stack) = seq.POSIXt(from = ISOdate(2016,1,17), by = "16 day", length.out = 23) # atribut name as date for each layer
my_palette = colorRampPalette(c("red", "yellow", "lightgreen")) #Create a color palette for our values
## Plot X maps in the same layout
spplot(ndvi.stack, layout=c(4, 6),
col.regions = my_palette(16))
Ce qui nous donne pour les 23 pas de temps étudiés l’image suivante. On voit bien reverdir la partie supérieure de la carte entre juillet et octobre en 2016.
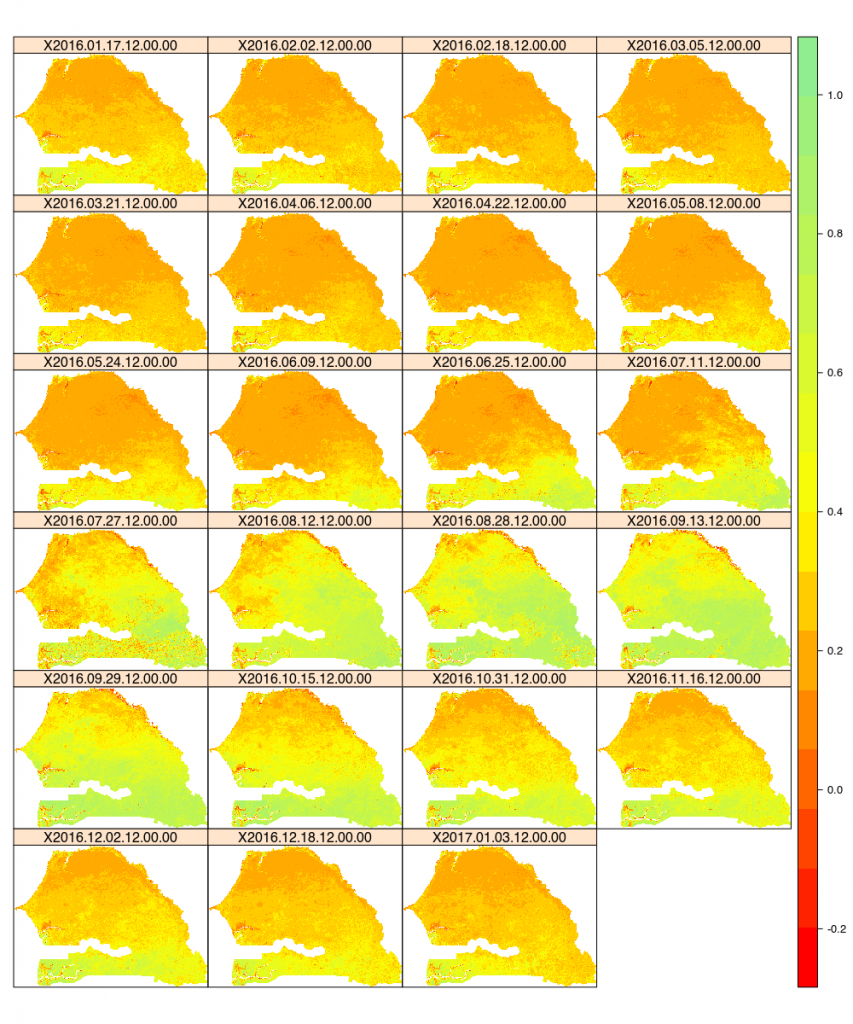
Pour avoir un ordre d’idée, traiter 23 images et produire le graphe, en parallélisant une partie, me prend 11 min .
Des ressources :
[1]Type d’images disponible : https://modis.gsfc.nasa.gov/data/dataprod/
[3]http://www.un-spider.org/sites/default/files/Skript%20VCI%20final.R