Du 19 au 24 juin 2023, j’ai pu participer au congrès de l’IASC (International Association for the Study of the Commons) qui s’est déroulé à Nairobi. La thématique de la conférence était « the commons we want« , a donné lieu à une très grande diversité de panels en parallèle et donc a un programme riche et varié.
J’ai eu la chance, presque un peu par hasard, de participer au workshop, puis au panel sur l’Institutional Gramar (IG) proposé par Saba Siddiki, Christopher Frantz, et Ute Brady. Ce qui m’a permis une introduction rapide aux potentialités de la formalisation proposée.
Une synthèse de l’institutional Grammar
L’approche de la Grammaire institutionnelle (Institutional Grammar ou IG en anglais) est une approche définie conceptuellement par Crawford et Ostrom en 1995. Au fil des 25 dernières années, cette approche a été largement utilisée dans l’analyse des politiques, la modélisation informatique, etc.
l’IG appelée « Institutional Grammar 2.0 », actualise l’approche pour de nouvelles applications dans divers domaines de recherche. Cette version 2.0 offre une représentation plus précise et complète des informations institutionnelles, en mettant l’accent sur le sens, les fonctions et les effets des déclarations institutionnelles.
La formalisation des informations en passant par un encodage particulier et non ambigu (ou en tout cas moins ambigu que le langage naturel), indépendamment du domaine d’application et de la méthode analytique utilisée, ouvre des perspectives extrêmement intéressantes et déjà largement utilisées en termes d’analyse comparée et d’évolution temporelle des organisations. En soulignant l’importance de l’analyse institutionnelle, il me semble que l’IG est adaptable à diverses situations d’actions.
La formalisation proposée par l’IG 2.0 repose sur une grammaire générative décrite dans la table ci dessous par Watkins et Westphal (2016).

L’intérêt de l’Institutional Grammar réside pour moi dans le cadre d’analyse rigoureux qu’elle offre et qui donne des outils holistiques pour étudier le fonctionnement des institutions et des organisations. En utilisant cette approche, les chercheurs peuvent mieux comprendre comment les règles, les normes et les valeurs institutionnelles influencent les comportements et les décisions des acteurs impliqués.
Cette compréhension approfondie des structures institutionnelles et de leurs effets permet de proposer des solutions plus adaptées pour résoudre les problèmes et les défis auxquels sont confrontées les organisations. L’Institutional Grammar peut également aider à identifier les facteurs qui favorisent ou entravent l’efficacité des institutions, ce qui peut avoir des implications importantes pour l’amélioration des politiques publiques et des pratiques organisationnelles.
De retour dans la vraie vie, j’ai essayé de comprendre plus finement la méthodologie et les usages que je pouvais en faire. J’ai identifier deux cas d’usage au Sénégal : autour du lac de Guiers dans le cadre du projet Santés & Territoires, et dans la zone pastorale dans le cadre du projet Dundi Ferlo, et dans le cadre de la convention de programme sur les Communs.
Santés & Territoires : une approche co-construite de one Health
Le projet s’appuie sur une approche participative, impliquant les acteurs du système autour de la transition agro-écologique comme levier d’amélioration des santés (approche one Health). Des chercheurs et des acteurs locaux sont donc impliqués dans 4 pays : au Sénégal, au Bénin, au Laos et au Cambodge, au travers de dispositifs de recherche-action que peuvent être les livings lab. Au Sénégal, les livings-lab se positionnent autour du lac de Guiers (à Mbane, et Keur Momar Sarr).
Entre février et septembre 2023, Amandine Adamczewski-Hertzog et moi avons encadré Mathilde Hibon dans le cadre de son stage de fin d’études pour un double diplôme entre l’école d’agronomie de Purpan et l’ISARA (Norwegian University of Life Sciences, vous pouvez retrouver son rapport ici).
Mathilde put encoder tous ses entretiens avec l’IG. Cela lui a donné 150 entrées dans sa base. À partir de là, on a exploré ces entrées avec des outils de l’analyse de réseau (R, et gephi). En les exprimant par point de vue. C’est à dire, envisager la manière dont chacun considère les relations d’interaction (au travers de règles ou de stratégies). Voilà le résultat de l’une de ces analyses du point de vue des populations locales.
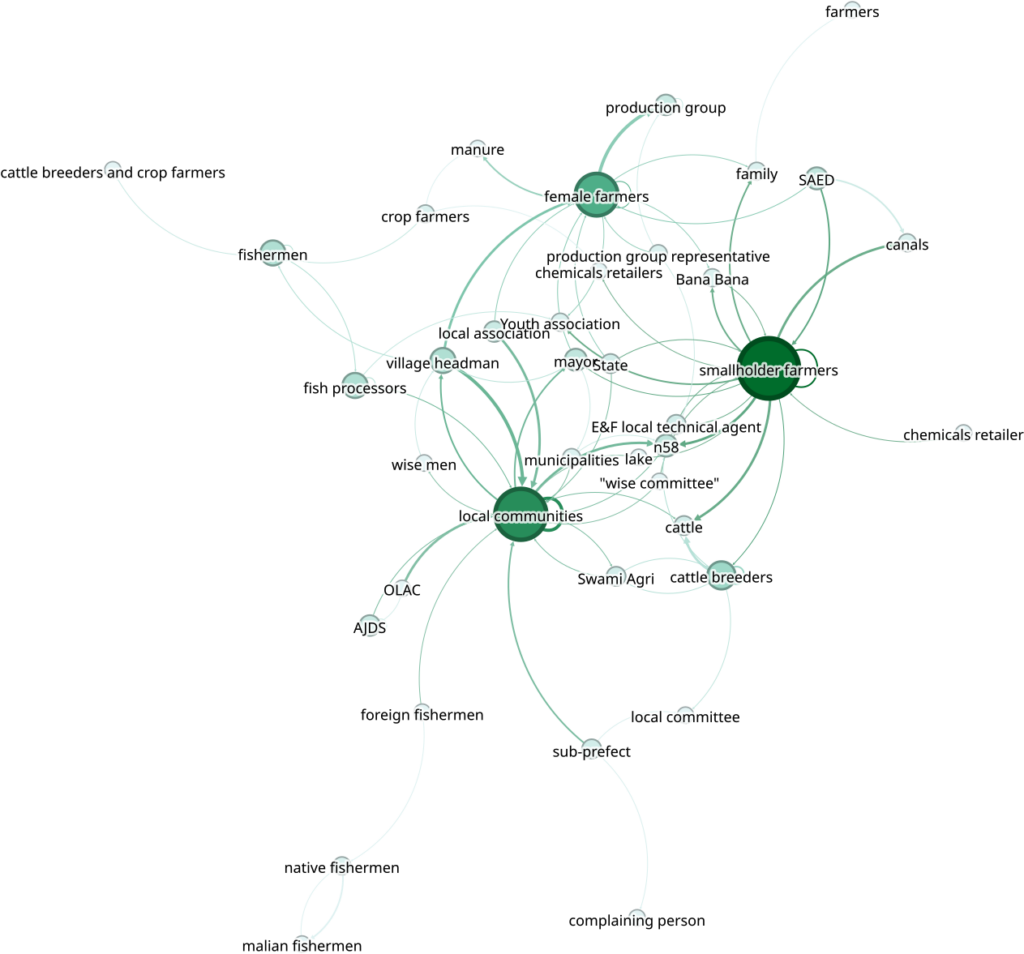
En regardant les relations qui sont identifiées à partir des entretiens mener auprès des institutions, on constate que la centralité des acteurs n’est pas du tout la même, ce qui permet entre autres choses de visualiser facilement les différences de points de vue.
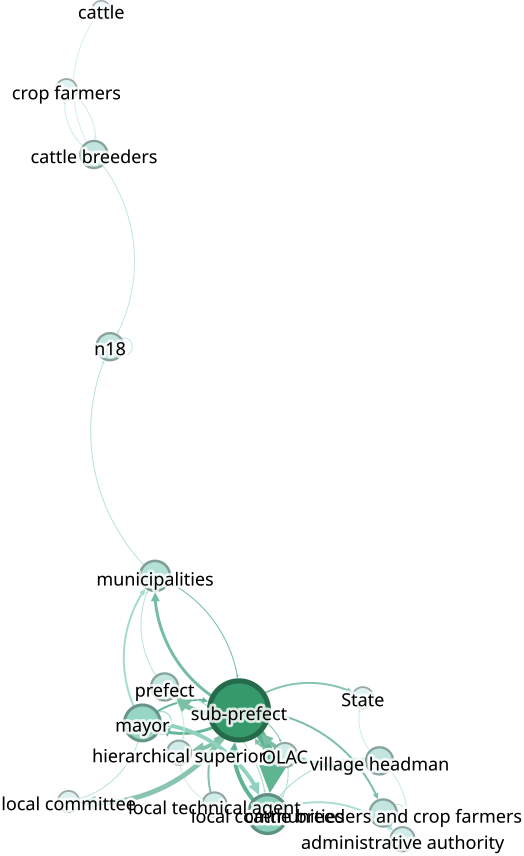
La CP Commun
Dans le cadre de la CP Commun, l’équipe GIREL du GRET Sénégal a séléctionné deux terrain : Mont-Rolland et Diender pour y déployer le jeu f’eau diem. Le jeu f’eau diem étant utilisé comme une arène qui permet de virtualiser les problèmes auxquels les participants font face. On réutilise à notre compte le concept de Johan Huizinga (1949) : “The Magic Circle”. Cela renvoie à l’espace conceptuel (mental) et physique (le lieu) dans lequel les règles du jeu prennent le dessus sur celles de la réalité quotidienne. L’identification de cet espace permet aux observateurs de la session de jeu de mieux comprendre les relations que les joueurs tissent localement avec la réalité. Comment ceux-ci adoptent des rôles ou des comportements qu’ils n’auraient pas dans leur vie quotidienne.

De cette manière nous nous attendons à ce que les joueurs ne nous parlent pas directement de leurs problèmes et de solutions de gestion mais de ceux identifiés dans le jeu, qui pour autant ont des équivalences dans la réalité.
A partir des observations des sessions de jeux, l’objectif est de remplir la matrice des formalisations des règles largement inspirées de l’Institutional gramar. Ce formalisme nous semblait être en mesure de capturer les éléments de réglementation formelle ou informelle que les participants allaient évoquer dans le cercle magique (c.f. fig I).
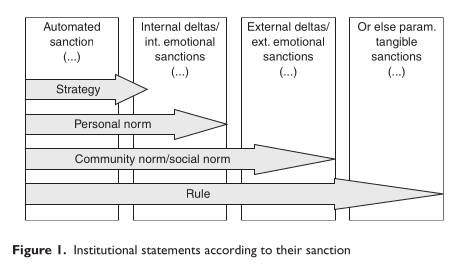
Les institutions (action d’instituer en économie) sont composées d’un ensemble de stratégies, normes et règles qui sont mises en place par un groupe d’agents. Pour un agent, une institution peut-être consciente et inconsciente. La Stratégie décrit le comportement d’un joueur dans un jeu. Elle peut être individuelle si on ne sait pas qui d’autre la mobilise. Collective, dans ce cas là, on assiste à l’apparition d’une institution. Les normes peuvent être internalisées, c’est-à-dire incorporées à l’intérieur de l’individu de manière à ce qu’il s’y conforme sans récompense ou punition externe, ou elles peuvent être imposées par des sanctions positives ou négatives venant de l’extérieur.
Les règles identifiées par les observateurs devaient être mises en discussion puis testées dans des sessions de jeux. S’il y a eu des adaptations locales, on peut globalement dire que le plan ne s’est pas passé comme prévu. Philippe Karpe (Cirad – UMR SENS) nous avait un peu mis en garde vis-a-vis de la grille. Elle était très compatible avec le droit positif, mais assez peu opérante pour faire face aux systèmes de régulation endogène mis en place par les participants. On a donc encore du travail 🙂
Dundi Ferlo : travailler avec les populations locales au projet de Grande Muraille Verte
Penda Diop Diallo, a emmené le CNRF (Centre National de Recherche Forestière Sénégalais) dans les approches de modélisation d’accompagnement (ComMod). Dans ce projet il s’agira de s’inscrire dans le projet panafricain de la grande muraille verte, en prenant en accompagnant les acteurs dans le processus. Dans les faits, cela veut dire accompagner la mise en place de parcelles de reboisement gérer collectivement par les populations locales aussi bien du point de vue du fourrage herbacé, que du potentiel fourrage aérien, et un jour des productions forestières non ligneuses.
Dans le cadre de ce travail, Penda accompagnée d’Anna Ndiaye ont réutilisé et adapté un jeu sérieux dans la plus pure approche ComMod. Il s’agissait de se servir de cet outil comme d’un simulateur social qui permettent aux acteurs de se projeter dans des mesures de gestion, d’en évaluer ainsi la faisabilité et les implications sur l’usage de la ressource.
Ces activités, réalisées à Vélingara Ferlo et Younofere, ont amené les acteurs à exprimer des ensembles de règles de gestion qui ont été implémentés la même année dans les parcelles. Penda a travaillé à la formalisation de ces règles dans l’Institutional Grammar pour pouvoir identifier les trajectoires institutionnelles que chacune des zones a décidé de prendre. Cela permettra de comparer facilement les directions que vont prendre les deux terrains d’étude.
Les perspectives
Si ça vous intéresse je vous encourage à aller voir le site de l’institutional Grammar initiative. En terme de perspective, il me semble que ce cadre est extrêmement stimulant, malgré les limites identifié précédemment, pour modéliser et donc simplifier les types de relation liés aux règles formelles et informelles dans la zone.