Dans le monde de la modélisation individu centré, l’un des formalismes attendus pour publier une description de modèles s’appelle ODD pour Overview, Design
concepts, and Details. La description de ce formalisme a été publiée en 2006 dans Ecological Modelling.
Un conteneur pour postgreSQL et postgis
Il arrive des moments dans la vie où on a besoin d’un petit serveur de base de données rapidement, là tout de suite. Si ça vous est déjà arrivé, vous savez que c’est parfois se lancer dans une aventure … qui si elle n’est pas périlleuse, peut s’avérer longue et douloureuse.

Alors, pourquoi ne pas en profiter pour passer à docker ? Parce qu’avec le principe de dockerfiles, l’installation ne prend pas plus longtemps que de télécharger les packages.
Installation docker sur Ubuntu
Source : docker.com
Le site de docker est très bien fait. Sur Ubuntu 16.04, il suffira de lancer les lignes de commandes suivantes.
sudo apt-get install -y --no-install-recommends apt-transport-https ca-certificates curl software-properties-common curl -fsSL https://apt.dockerproject.org/gpg | sudo apt-key add - apt-key fingerprint 58118E89F3A912897C070ADBF76221572C52609D sudo add-apt-repository "deb https://apt.dockerproject.org/repo/ \ ubuntu-$(lsb_release -cs) \ main" sudo apt-get update sudo apt-get -y install docker-ce
Si tout s’est déroulé sans accrocs, vous pouvez vérifier que docker fonctionne avec :
sudo docker run hello-world
Il ne reste enfin quelques petits ajustements à faire, notamment configurer votre utilisateur dans le groupe docker pour s’abstraire de l’utilisation de sudo par exemple. Vous trouverez ces choses-là sur cette page.
Installation de postGIS et postgreSQL
Source : postgis dockerfile
Pour créer un conteneur docker il suffit de lancer la commande docker run. Docker va vérifier si vous disposez d’une « image » du service que vous souhaitez lancer. Si c’est le cas, un conteneur sera créé, sinon les différentes composantes de l’image seront téléchargées et le conteneur sera créé dans la foulée.
On commence ici par créer un conteneur postGIS qui s’appellera psql-futurSahel (C’est l’étiquette du conteneur et cela n’aura pas d’influence sur la base de données). On définit le mot de passe de postgreSQL et on spécifie que l’on veut utiliser l’image postgis fournie par l’utilisateur mdillon.
docker run --name psql-futurSahel -e POSTGRES_PASSWORD=postgres -d -p 5432:5432 mdillon/postgis
Pour que cela fonction, il faut également installer un postgreSQL et le lier à l’image de postGIS. Ce qui nous permettra ensuite d’accéder à l’interface de base de données et de nous connecter avec psql
docker run -it --link psql-futurSahel:postgres --rm postgres \ sh -c 'exec psql -h "$POSTGRES_PORT_5432_TCP_ADDR" -p "$POSTGRES_PORT_5432_TCP_PORT" -U postgres'
Dans Postgre
Si tout s’est passé comme il faut, suite à la dernière commande, et après téléchargement des différents constituants de l’image, vous êtes rentré dans le conteneur et vous avez en face de vous un prompt psql.
Vous pouvez donc utiliser les commandes habituelles de postgre. Il faut, une fois connecté à psql, créer une base de données
CREATE DATABASE fs_gis;
On peut ensuite se connecter à la base de données
\connect fs_gis;
Et lancer la commande
CREATE EXTENSION postgis;
La base est donc créée en utilisant le template postGIS.
Peupler la base de données
Vous avez maintenant un serveur de base de données spatiales qui tourne et auquel vous pouvez accéder depuis la machine hôte.
Pour connaître l’IP du conteneur :
docker inspect idconteneur
Vous pouvez alors configurer vos outils de gestion de base de données en utilisant l’IP du conteneur et/ou votre propre IP (ifconfig).
Dans Qgis vous pouvez configurer la gestion de la base de la manière suivante :
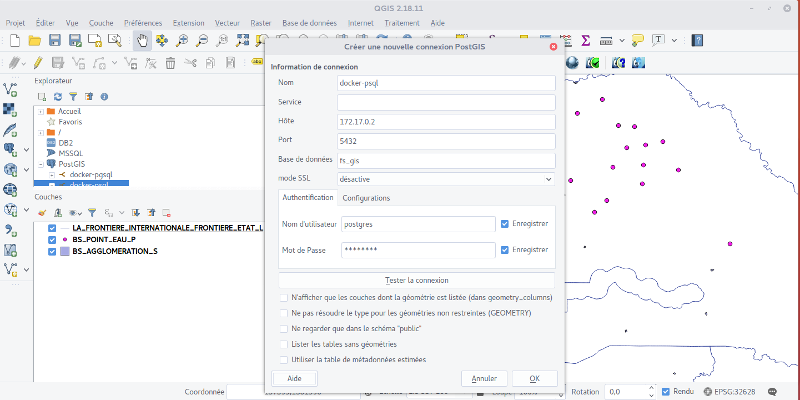
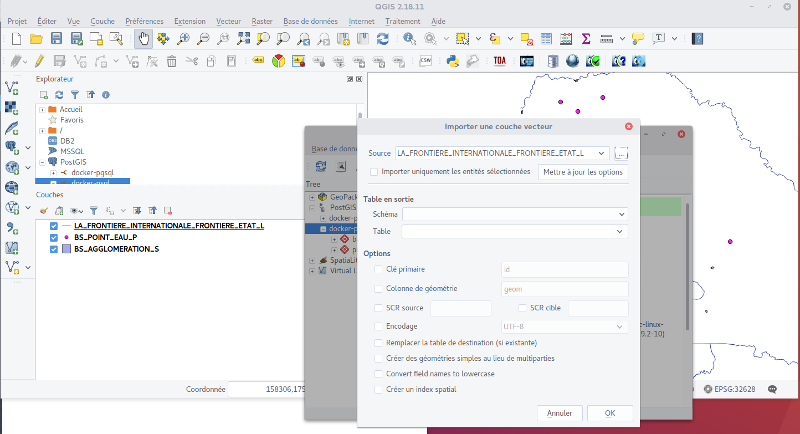
Et vous pouvez utiliser l’extension Base de données > Gestionnaire de base de données > Gestionnaire de base de données, pour importer dans postgreSQL vos données en cliquant sur ‘importer une couche ou un fichier’. Cet outil va utiliser le script shp2psql de manière transparente pour l’utilisateur.
Traiter des images MODIS avec R pour calculer un NDVI
La Nasa propose depuis 1999 des images MODIS (Moderate-Resolution Imaging Spectroradiometer) à différentes résolutions spatiales et temporelles. Elles sont produites par deux satellites en orbite, Terra (1999) et Aqua (2002), qui embarquent des capteurs pour le programme Earth Observing system.
Les instruments permettent d’enregistrer 36 bandes spectrales avec une fréquence de passage de 1 à 2 jours. La résolution des images diffère selon les bandes enregistrées et varie entre 0.25 et 1 km. Nous parlions il y a quelques jours du site reverb|echo de la Nasa, il se trouve qu’un certain nombre d’images sont disponibles sur le site.
Quelles données ?
Mais pour ça il faut savoir ce qu’on cherche, et la nomenclature des produits n’est pas vraiment transparente [1]. À titre d’exemple :
- MOD9Q1 – Surface Reflectance 8-Day L3 Global 250m
- MOD13Q1 – Vegetation Indices 16-Day L3 Global 250m
- MOD11A2 – Land Surface Temperature/Emissivity 8-Day L3 Global 1km
Une fois qu’on en sait un peu plus sur le type de produit que l’on veut utiliser, il va falloir le télécharger et le traiter. Dans un post précédent, j’expliquais comment configurer sa machine pour utiliser wget. Si vous optez pour cette solution, il vous faudra utiliser le MRT pour reprojeter et assembler vos images. L’interface est assez intuitive et on peut faire du traitement de masse.
Mais je suis tombé aussi ce matin sur les logs de release d’un package R que je me suis empressé de tester : MODIStsp. Si vous avez déjà regardé MRT, MODISStsp n’est qu’un moyen de rester dans R pour télécharger les images. On pourra aussi regarder du côté du package MODIS qui lui n’intègre pas d’interface graphique.
Bon mais on parlait de NDVI dans le titre !
Traiter les images NDVI : MOD13Q1
En cherchant comment traiter les images MODIS, je suis tombé sur des ressources intéressantes. Notamment une page qui vous prend par la main pour traiter les données MODIS (MOD13Q1) dans R[2]. Je vous propose donc de faire un petit point pas à pas !
J’ai téléchargé et projeté les données grâce au package MODISStsp. J’ai donc un dossier qui contient les fichiers hrf, mais aussi un autre qui contient les fichiers tiff produits par le package.
Or quand on ouvre les données (dans Qgis), les gammes de valeurs ne correspondent pas à celles attendues pour du NDVI! On devrait avoir des valeurs entre -1 et 1 !
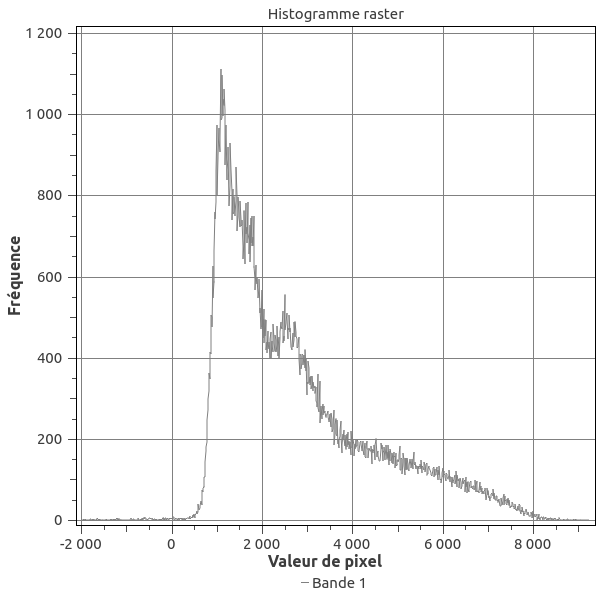
Il va falloir retravailler le raster pour obtenir les bonnes valeurs . Heureusement « the office of outer space affaire » nous donne la marche à suivre dans R[3]. Je vous propose donc ici un script commenté et customisé pour l’occasion.
library(raster)
library(sp)
library(doParallel)
library(rgdal)
library(magrittr)
rm(list = ls()) ##clear env.
#create stack from tif NDVI files
path.v = "~/Documents/futurSahel/MOD13Q1_TS/VI_16Days_250m_v6/NDVI/"
l.files = list.files(path.v, pattern = ".tif")
my.stack = stack(paste0(path.v,l.files)) ##create raster stack of files in a directory
# load borders
border = shapefile("~/Documents/futurSahel/Senegal_gadm.org/SEN_adm0.shp") #ToDo: insert link to the shapefile with the country borders
## We use doParallel and magrittr packages to pipe different actions (crop and mask)
registerDoParallel(6) #we will use 6 parrallel thread
result = foreach(i = 1:dim(my.stack)[3],.packages='raster',.inorder=T) %dopar% {
my.stack[[i]] %>%
crop(border) %>%
mask(border)
}
endCluster()
ndvi.stack = stack(result)
ndvi.stack = ndvi.stack*0.0001 #rescaling of MODIS data
ndvi.stack[ndvi.stack ==-0.3]=NA #Fill value(-0,3) in NA
ndvi.stack[ndvi.stack<(-0.2)]=NA # as valid range is -0.2 -1 , all values smaller than -0,2 are masked out
names(ndvi.stack) = seq.POSIXt(from = ISOdate(2016,1,17), by = "16 day", length.out = 23) # atribut name as date for each layer
my_palette = colorRampPalette(c("red", "yellow", "lightgreen")) #Create a color palette for our values
## Plot X maps in the same layout
spplot(ndvi.stack, layout=c(4, 6),
col.regions = my_palette(16))
Ce qui nous donne pour les 23 pas de temps étudiés l’image suivante. On voit bien reverdir la partie supérieure de la carte entre juillet et octobre en 2016.
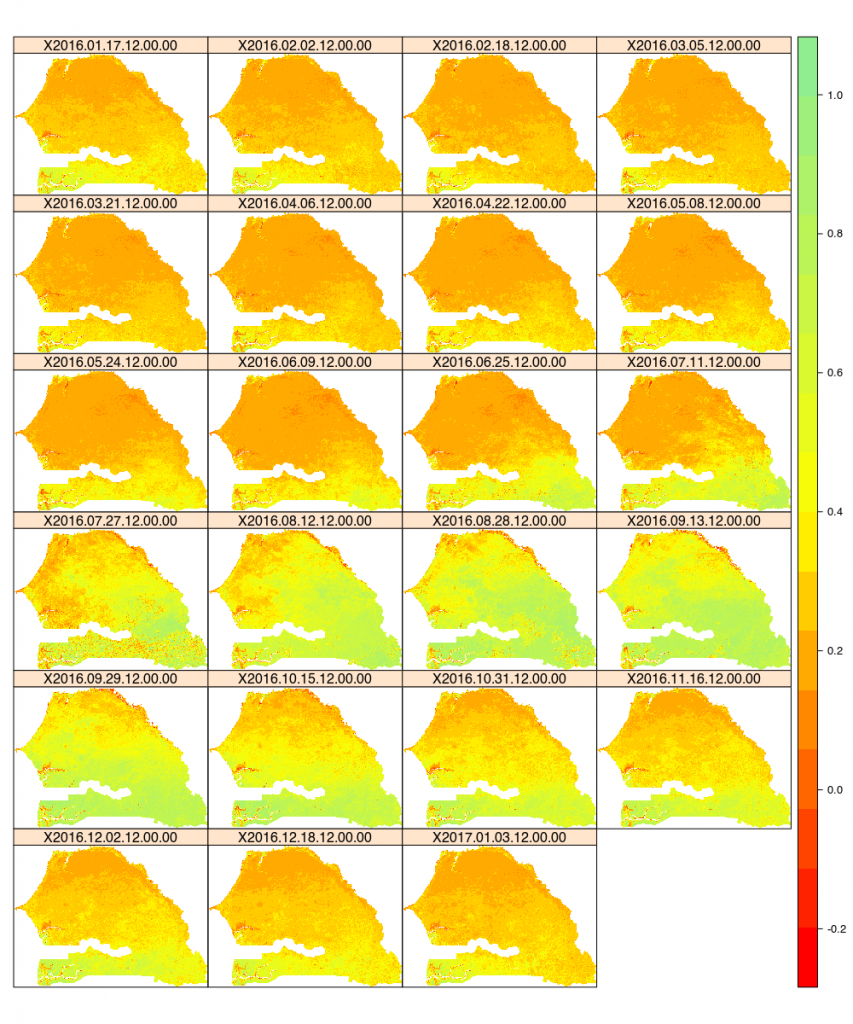
Pour avoir un ordre d’idée, traiter 23 images et produire le graphe, en parallélisant une partie, me prend 11 min .
Des ressources :
[1]Type d’images disponible : https://modis.gsfc.nasa.gov/data/dataprod/
[3]http://www.un-spider.org/sites/default/files/Skript%20VCI%20final.R
Earthdata des données SIG pour l’environnement
Voilà plusieurs mois que je n’avais pas pris le temps de faire un post sur mon blog !
On trouve toujours beaucoup d’excuses, des papiers sur le feu, un déménagement, un nouveau post-doctorat… Alors pour recommencer doucement, une petite note sur un service de la NASA que j’ai découvert aujourd’hui : Reverb | echo[1]

Un peu de contexte à cette découverte, mon nouveau post-doc m’a amené à sortir de ma zone de confort, on s’éloigne de la viticulture et de l’eau (encore que), pour travailler sur la zone sahélienne du Sénégal. Je cherchais des indicateurs de désertification… Et je suis tombé sur un blog génial de Martin Brandt[2] (chercheur au Department of Geosciences and Natural Resource Management à l’université de Copenhagen (Denmark)). En plus de pointer sur des papiers scientifiques, il y a aussi quelques posts sur des outils de télédétection (GRASS-GIS) et des liens vers des pourvoyeurs de données. C’est là que j’ai découvert LE truc.
Reverb est un silo de données environnementales propulsées par la NASA qui contient des choses incroyables et un puissant moteur de recherche. Je vous engage donc à regarder cette petite vidéo d’explication[3] (en anglais).
Je ne reviendrai pas sur la manière de trouver ses données, la vidéo est là, par contre une chose géniale qui pourrait inspirer d’autres sites : une fois que j’ai fait mon petit marché, le service me propose de télécharger un fichier TXT contenant les URLs des ressources demandées. L’idée est génial. Il y a quelques semaines, j’avais du faire une centaine de copier-coller depuis le site opendata.gouv.fr pour pouvoir automatiser un traitement sur le RGP de 2012[4] (j’en parlerai peut-être dans quelques mois quand l’article sera publié).
Donc une grande satisfaction, je disais, pour cette bonne pratique… je voulais donc me lancer dans un script bash et du wget… mais les ennuis arrivent …
wget http://e4ftl01.cr.usgs.gov//MODV6_Cmp_B/MOLT/MOD09Q1.006/2016.09.05/MOD09Q1.A2016249.h16v07.006.2016258071050.hdf Connexion à urs.earthdata.nasa.gov (urs.earthdata.nasa.gov)|198.118.243.33|:443… connecté. requête HTTP transmise, en attente de la réponse… 401 Unauthorized Échec d’authentification par identifiant et mot de passe. URL transformed to HTTPS due to an HSTS policy
Mince un login ! Nooon ! Pourtant Martin Brandt ne semble pas avoir ce problème. La solution est en deux étapes. Dans un premier temps, il faudra se créer un compte « earthdata » en cliquant sur login en haut à gauche. Ensuite, et la solution est sur le wiki [5], il faudra faire un petit fichier de config « curl ».
cd ~ touch .netrc echo "machine urs.earthdata.nasa.gov login; password " > .netrc chmod 0600 .netrc
Vous l’aurez compris, il ne reste qu’à remplacer
wget http://e4ftl01.cr.usgs.gov//MODV6_Cmp_B/MOLT/MOD09Q1.006/2016.09.05/MOD09Q1.A2016249.h16v07.006.2016258071050.hdf Sauvegarde en : « MOD09Q1.A2016249.h16v07.006.2016258071050.hdf » MOD09Q1.A2016249.h1 100%[===================>] 64,76M 344KB/s in 1m 51s
Merci pour les données, et merci pour cette facilité d’utilisation !!!

Les liens
[1]https://reverb.echo.nasa.gov
[2]https://matinbrandt.wordpress.com
[3]https://www.youtube.com/watch?v=iBXzzUv3b4w
[4]https://www.data.gouv.fr/fr/datasets/registre-parcellaire-graphique-2012-contours-des-ilots-culturaux-et-leur-groupe-de-cultures-majorita/
[5]https://wiki.earthdata.nasa.gov/display/EL/How+To+Access+Data+With+cURL+And+Wget
R et Netlogo sont sur un bateau : OpenMole
J’ai participé en juillet au coding-camp OpenMole (j’y reviendrais surement plus tard, ou pas), où j’ai découvert un moyen d’embarquer R dans OpenMole ! Cela m’ouvre des perspectives inimaginables il y a encore quelques semaines !

Et oui parce qu’on va pouvoir piloter Netlogo avec R dans OpenMole ! Un vrai jeu de poupée russe ! Mais comme les poupées russes, il va falloir procéder par étape pour pouvoir bénéficier de la puissance de la parallélisation sur son propre desktop comme sur un cluster ou une grille et cela sans effort supplémentaire !
Etape 1 : Netlogo dans R
Je vais sauter celle du modèle Netlogo fonctionnel, pour l’exemple on va prendre un modèle de la librairie : ants . La première chose qu’on va faire c’est l’embarquer dans R. Pour cela il faudra avoir le package RNetlogo installé évidemment .
##Script R pour explorer les données le modèle ants de NetLogo
#Chargement du package RNetLogo
library(RNetLogo)
#localiser l'installation de netlogo
nl.path = "/opt/netlogo-5.3.1-64/app/"
NLStart(nl.path, gui = TRUE) #lance netlogo avec (TRUE) ou non (FALSE) une GUI
##Définition du chemin du modèle
model.path = "/opt/netlogo-5.3.1-64/app/models/Sample Models/Biology/Ants.nlogo"
## chargement du modèle dans netlogo
NLLoadModel(model.path)
#################################
## CREATION DU PLAN D'EXPERIENCE
#################################
# Ici on va définir le domaine d'exploration de notre modèle
# Pour simplifier ici on le met dans le même script, mais il serait plus
# judicieux de l'écrire dans un fichier Rdata ou csv quand on passera à
# l'encapsulage dans OpenMole
diff.rate = seq(from = 0, to = 100, by = 10) ##explorer le parametre de 0 à 100 par pas de 10
evap.rate = seq(from = 0, to = 100, by = 10)
pop.dif = seq(from = 40, to = 200, by = 20)
replication = seq(from = 1, to = 10, by = 1)
#Création d'un data frame avec toutes les combinaison des paramètres
pl.exp = expand.grid(pop = pop.dif, diff = diff.rate, evap = evap.rate)
#################################
## RUN Netlogo
#################################
##créer un dataframe vide qui sera supprimé quand on passera à openmole
data.df = data.frame()
#for(i in 1:length(pl.exp[,1])){
for(i in 1:5){
# Pour chaque ligne du tableau on va lancer une simulation
# ATTENTION : Cette première boucle se fera par la suite dans OpenMole
##Maping des variables dans netlogo
NLCommand("set population ", pl.exp$pop[i])
NLCommand("set diffusion-rate ", pl.exp$diff[i])
NLCommand("set evaporation-rate ", pl.exp$evap[i])
## Lancement de la commande setup du modèle
NLCommand("setup")
## créer un reporter qui stock la valeur de la somme de nourriture des patches
food = NLDoReport(2000, "go", "sum [food] of patches")
data.df = cbind(data.df, food)
}
data.df = t(data.df)
plot(x = 1:length(data.df[,1]), y = data.df[,2], type = "l")
On a maintenant un script fonctionnel, mais il faudrait pouvoir le paralléliser. Si on voulait rester dans R il y aurait des solutions assez faciles avec les packages « snow » ou « parallel ». Cela nous conduirait à paralléliser pour une machine donnée. En utilisant OpenMole, on va être capable de paralléliser de manière transparente sur notre ordinateur de bureau, mais aussi sans effort et avec un script identique sur un cluster ou une grille de calcul.
Etape 2 : empaqueter R avec care
Voilà l’étape un peu fastidieuse (à mon sens) mais nécessaire. Les dev. d’OpenMole nous invite à utiliser care, un logiciel qui va scanner notre script pour créer une archive contenant toutes les dépendances dont aura besoin openMole pour exécuter une instance.
Modifier le srcipt
Avant cela une petite modification du script R s’impose . L’idée ici est de passer un argument au script pour lui spécifier la ligne du jeu de paramètres que l’on veut lancer et stocker les résultats dans un fichier CSV (par exemple).
##Script R pour explorer les données le modèle ants de NetLogo
# Notre script va accepter un argument passé en ligne de commande
args<-commandArgs(trailingOnly = TRUE)
args <- as.numeric(args) ## on passe un chiffre
#Chargement du package RNetLogo sans message d'info
suppressPackageStartupMessages(library(RNetLogo, quietly = TRUE, warn.conflicts = TRUE))
#localiser l'installation de netlogo
nl.path = "/opt/netlogo-5.3.1-64/app/"
NLStart(nl.path, gui = FALSE) #lance netlogo avec (TRUE) ou non (FALSE) une gui
##Définition du chemin du modèle
model.path = "/opt/netlogo-5.3.1-64/app/models/Sample Models/Biology/Ants.nlogo"
## chargement du modelé dans netlogo
NLLoadModel(model.path)
#################################
## CREATION DU PLAN D'EXPERIENCE
#################################
# Ici on va définir le domaine d'exploration de notre modèle
# Pour simplifier ici on le met dans le même script, mais il serait plus
# judicieux de l'écrire dans un fichier Rdata ou csv quand on passera à
# l'encapsulage dans OpenMole
diff.rate = seq(from = 0, to = 100, by = 10) ##explorer le parametre de 0 à 100 par pas de 10
evap.rate = seq(from = 0, to = 100, by = 10)
pop.dif = seq(from = 40, to = 200, by = 20)
replication = seq(from = 1, to = 10, by = 1)
#Creation d'un data frame avec toutes les combinaisons des paramètres
pl.exp = expand.grid(pop = pop.dif, diff = diff.rate, evap = evap.rate)
#################################
## RUN Netlogo
#################################
##Maping des variables dans netlogo
NLCommand("set population ", pl.exp$pop[args])
NLCommand("set diffusion-rate ", pl.exp$diff[args])
NLCommand("set evaporation-rate ", pl.exp$evap[args])
## Lancement de la commande setup du modele
NLCommand("setup")
## creer un reporter qui stock la valeur de la somme de nourriture des patches
food = as.data.frame(NLDoReport(2000, "go", "sum [food] of patches"))
#
#
save(food, file = "/mypath/food.RData")
NLQuit()
Une fois le script modifié, on peut tester que tout fonctionne
R --slave -f run_netlogo.R --args 4
Ce qui doit avoir pour effet de sauvegarder un fichier food.Rdata en itilisant la ligne 4 (args) du data frame créé dans le script.
Créer un package pour OpenMole
L’idée ici est de créer un package qui contient R, le script, et toutes ses dépendances (RNetlogo et Netlogo). Pour cela on utilisera care. Il vous faudra télécharger l’exécutable et le rendre exécutable soit dans le répertoire courant, soit dans les bins.
Ensuite l’usage est assez simple :
./care-x86_64 -o r.tar.gz.bin R --slave -f run_netlogo.R --args 4
Cela va avoir pour effet de lancer dans care votre script et lui permettre de détecter les dépendances. Cela peut prendre du temps en fonction du temps d’exécution de votre script.
Vous obtenez en sortie une archive nommée r.tar.gz.bin et vous pourrez passer à OpenMole !
Etape 3 : Embarquer R dans OpenMole
Vous pouvez lancer votre session OpenMole.
cd openmole_ex_dir/ ./openmole
Cela devrait avoir pour effet d’ouvrir votre navigateur préféré : Firefox (ou chrome… ça marche aussi). Dans l’interface graphique, vous pouvez charger une archive care.

Un widget bien fait détecte le paramétrage de votre script et vous propose un squelette de script openmole. Il ne vous restera plus qu’à le peaufiner pour pouvoir paralléliser tout ça. 😀
val i3 = Val[Int] //table of correspondance
val output = Val[File]
val rTask = CARETask(workDirectory / "myscript.tar.gz.bin", "R --slave -f run_grid.R --args ${i3}") set(
inputs += (i3),
//Default values. Can be removed if OpenMOLE Vals are set by values coming from the workflow
outputs += (i3),
outputFiles += ("simulation.RData", output)
)
val copyHook = CopyFileHook(output, workDirectory / "resultats/run_result_${i3}.RData")
val exploration = ExplorationTask(
(i3 in (1 to 24 by 1))
)
val env = LocalEnvironment(2) //defined number of thread used by the mole
exploration -< (rTask hook copyHook on env)
Enjoy et merci pour le travail incroyable de la team OpenMole !
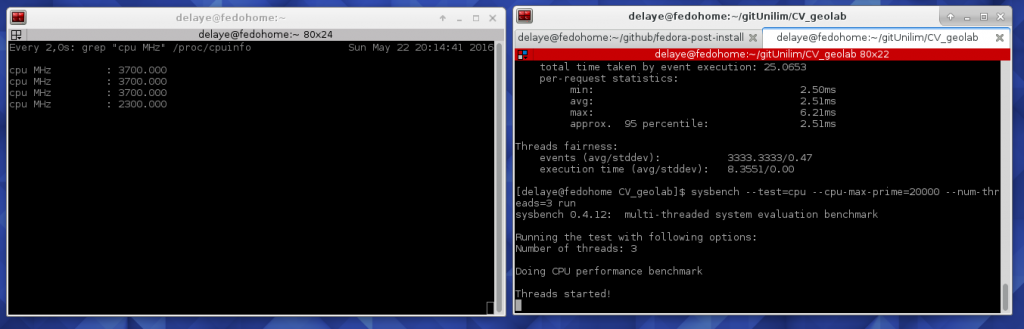
benchmark CPU avec linux
Un vieux portable qui craque, une installation Gnu-Linux toute neuve ! Et voilà que me vient l’envie de regarder comment fonctionne mon CPU histoire de voir si je n’ai pas un thread qui a grillé :-p ! J’ai donc cherché à droite et à gauche des solutions en ligne de commande ! Et voilà! On ouvre deux terminaux et dans chacun on colle l’une des lignes suivantes :
La première va lancer, sur 4 thread 2000 calcule
sysbench --test=cpu --cpu-max-prime=20000 --num-threads=4 run
La seconde va nous permettre d’observer l’évolution de la fréquence du CPU
watch grep \"cpu MHz\" /proc/cpuinfo
Et voilà on peut voir 4 thread entre 800 et 3700 MHz ! Tout fonctionne 🙂
GRASS-GIS 7 et R
Contexte
Avec @fabio_zottele, nous nous sommes replongés ce weekend sur un travail un peu vieux. Celui-ci remonte à 5 ans, et consiste à proposer un algorithme basé sur des librairies/logiciels libres afin de détecter les terrasses viticoles. Ce travail avait initialement été développé sur R gdal et grass 6.3. pour une petite zone de la val di Cembra.
Notre objectif est aujourd’hui de systématiser l’algorithme et de vérifier que l’évolution des technologies et des algorithmes sur lesquels nous nous étions basés produit toujours le résultat attendu.
Installation de GRASS7
La première chose à faire est de compiler GRASS-GIS en version 7, car les dépôts ne sont pas encore à jour sur Fedora. Vous pouvez trouver toute la documentation sur le wiki de grass.
On pourra commencer par télécharger le weekly snapshot du projet , et installer les dépendances qui vont bien :
sudo dnf install gcc gcc-c++ bison flex ncurses-devel proj-epsg proj-nad xml2 \ proj-devel gdal gdal-devel gdal-python sqlite-devel mesa-libGL-devel \ fftw-devel mesa-libGLU-devel libXmu-devel libX11-devel geos geos-devel \ libtiff-devel lesstif-devel python-devel numpy wxPython wxGTK-devel \ python-dateutil python-imaging python-matplotlib-wx subversion doxygen python-sphinx \ netcdf-devel postgresql-devel lapack lapack-devel blas blas-devel atlas atlas-devel
On peut ensuite aller compiler les sources :
cd ~/Téléchargement/grass7.0.svn/ ./configure \ --with-cxx \ --with-gdal=/usr/bin/gdal-config \ --with-proj --with-proj-share=/usr/share/proj \ --with-python=/usr/bin/python-config \ --with-geos \ --with-sqlite \ --with-nls \ --with-wxwidgets=/usr/bin/wx-config \ --with-fftw \ --with-cairo --with-cairo-ldflags=-lfontconfig \ --with-freetype --with-freetype-includes=/usr/include/freetype2 \ --enable-largefile \ --without-odbc \ --with-ffmpeg \ --with-ffmpeg-includes="/usr/include/ffmpeg /usr/include/ffmpeg/libav* /usr/include/ffmpeg/libpostproc /usr/include/ffmpeg/libswscale" \ --with-blas --with-blas-includes=/usr/include/atlas-x86_64-base/ \ --with-lapack --with-lapack-includes=/usr/include/atlas-x86_64-base/ \ make -j4 #pour compiler sur 4 thread sudo make install
Et nous voilà avec une belle installation de grass7. Je vous encourage à faire un tour du propriétaire parce qu’un travail formidable a été fait ! Bravo à l’équipe de dev!
Comment se passe la communication avec R ?
 Et bien figurez-vous qu’il y a le package qui va bien 😀 : rgrass7 qui s’installe comme d’habitude dans R avec :
Et bien figurez-vous qu’il y a le package qui va bien 😀 : rgrass7 qui s’installe comme d’habitude dans R avec :
install.packages(« rgrass7 », dependences = TRUE)
Mais l’utilisation diffère un peu de son grand frère. En effet, on peut désormais spécifier l’emplacement de votre GRASS7 fraichement compilé. On procèdera donc de la manière suivante :
remove(list=ls()) ## Configuration Parametrs: gisBaseLocation = "/opt/grass" #where the GRASS executables are library(rgrass7) #load lib in R initGRASS(gisBase=gisBaseLocation, override=TRUE) #Run GRASS in & Cancel previous running GRASS instance in R
L’exécution se fait ensuite avec une seule et même commande R à laquelle on passera en paramètre la commande GRASS
execGRASS(cmd= "g.remove", flags=c("f", "b"), parameters= list(type="raster", pattern="*"))
Ici on exécute donc dans R une commande GRASS qui va permettre de supprimer tous les rasters de la base de données GRASS.
Pour prendre un autre exemple :
execGRASS(cmd= "r.param.scale", flags= c("overwrite", "verbose"), parameters= list(input="DTM",output="CURVATURE", size=5, method="profc", zscale=2.0))
Ici on exécutera la commande r.param.scale en spécifiant toutes les options.
Quand on veut ensuite sortir les résultats de GRASS pour les manipuler dans R, la méthode n’a pas beaucoup changé :
param.scale = raster(readRAST("CURVATURE"))
Ici on utilise la fonction raster du package éponyme pour convertir en raster les données en sortie de la fonction readRAST du package rgrass7.
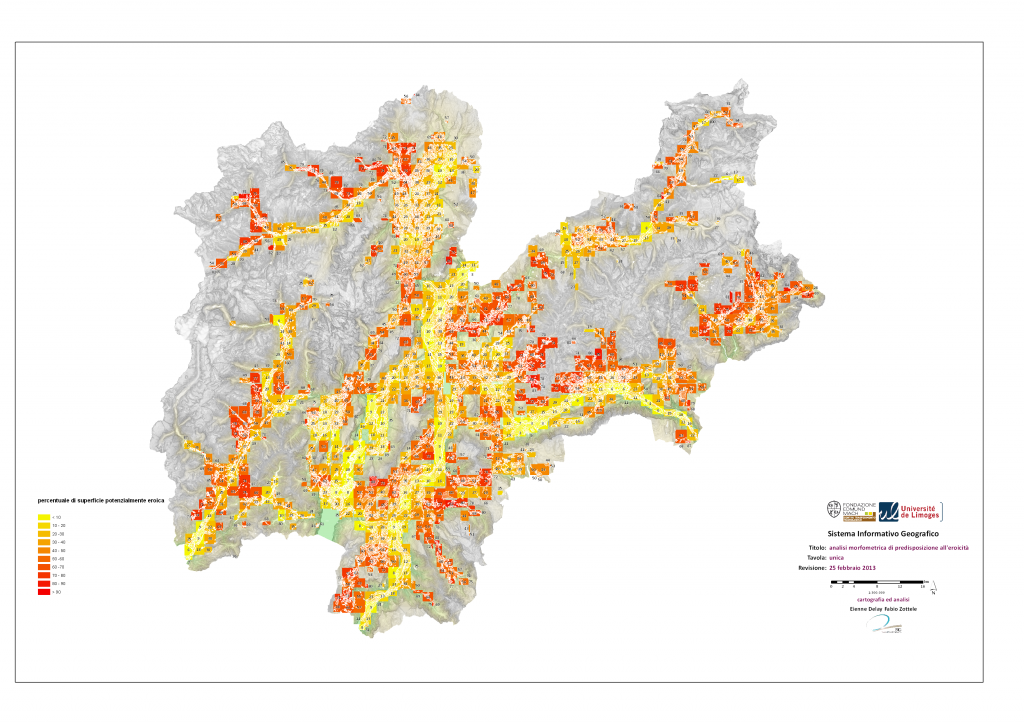
Pour le plaisir des yeux
Voilà donc à partir de données LIDAR à 1m la proportion de surface avec des terrasses à l’échelle tu Trentino !
Gephi 0.9 et Fedora 22
Les développeurs de Gephi nous ont fait un beau cadeau de Noël : la sortie de la version 0.9 de gephi. Ce qui me donne une bonne raison de vouloir le tester ce matin.
Je me suis donc lancé dans l’installation la fleur au fusil. Une fois le téléchargement effectué et la décompression faite, je lance fébrile le logiciel… gephi 0.8 n’avait jamais fonctionné de manière satisfaisante sur ma machine (Fedora 22 en 64 bits).
Caramba encore raté ! 
./gephi
me renvoie des erreurs, et si le logiciel se lance, je ne peux pas charger mon fichier gexf.
libEGL warning: DRI2: failed to open r600 (search paths /usr/lib/dri) libEGL warning: DRI2: failed to open swrast (search paths /usr/lib/dri) libGL error: unable to load driver: r600_dri.so libGL error: driver pointer missing libGL error: failed to load driver: r600 libGL error: unable to load driver: swrast_dri.so libGL error: failed to load driver: swrast libEGL warning: DRI2: failed to open r600 (search paths /usr/lib/dri) libEGL warning: DRI2: failed to open swrast (search paths /usr/lib/dri)
Pourtant j’ai bien la librairie LibEGL ! Quelques visites de forum plus tard, il semble que le même problème se présente avec l’utilisation de steam sur des architectures 64bits. Et que le problème est résolut par l’installation du package mesa-dri-drivers.i686.
#dnf install mesa-dri-drivers.i686
et voilà gephi qui charge mon fichier ! Pas plus de difficulté ! Et en plus je n’ai pas installé Java proposé par Oracle, mais openJDK ! En tous cas merci la Team de dev gephi pour ce beau travail !
passage si vous cherchez quelques jeux de données super : la page de gephi dédiée, mais aussi quelques jeux de données bien geek dans l’univers de Marvel proposé par Kai Chang, Tom Turner, et Jefferson Braswell
Hyphe un web crawler pour l’HNA !
Je travaille depuis une semaine pour la Chaire « Capital Environnemental et la Gestion Durable des Cours d’Eau » de la fondation universitaire de Limoges. L’objectif est de travailler sur la dimension collective de gestion de l’eau et plus particulièrement sur les formes sociales qu’elle peuvent prendre.
La première chose que j’ai voulu faire a été de crawler un peu de web pour espérer comprendre comment étaient structurées les organisations autour de la gestion de l’eau sur mon terrain.
Pour cela, j’ai cherché pendant plusieurs heures un outil qui puisse, à partir d’une URL, identifier les liens et retrouver la constellation des renvois que font les sites institutionnels entre eux ; ce que les anglo-saxon appelle le HNA (hyperlink network analysis).
Si ma première réaction a été de me tourner vers R, je n’ai pas vraiment trouvé mon bonheur, et je ne voulais pas ré-écrire un web crawler. Du coup mes explorations m’ont poussé à découvrir :
- SocNetv (un logiciel de réseau en Qt)
- voson (un webservice sur inscription)
- hyphe (un logiciel libre basé sur python et mongoDB)
Il se trouve que j’ai particulièrement aimé ce que propose hyphe, mais il n’est pas (encore) à proprement parlé multi-plateforme. Je me suis dit que ce serait l’occasion de tester Docker qui est une solution de virtualisation d’applications.
Docker quezako
Pour avoir un bon aperçu de ce qu’est Docker pour la virtualisation je vous encourage à lire un très bon post sur le blog de neogeo.
Utiliser Docker c’est magique ! Pour créer un conteneur sous Ubuntu voilà la marche à suivre.
sudo docker run -i -t ubuntu /bin/bash adduser delaye apt-get install sudo wget nano nano /etc/sudoers nano /usr/sbin/policy-rc.d exit 0 #à la place de 101
Vous avez alors accès à une machine dans laquelle vous allez pouvoir monter un serveur, une application ou tout autre chose qui peut servir dans la vie d’un chercheur. Mais encore plus fort, vous pourrez déplacer le tout comme un fichier. Ce qui veut dire que vous pourrez échanger avec d’autres vos applications fonctionnelles dans des conteneurs.
Docker + Hyphe = <3
Revenons à nos moutons, la beauté du couple Docker / Hyphe, c’est que vous disposez sur le github de Hyphe d’un Dockerfile. Le Dockerfile vous permet dans un seul ficher texte de définir la configuration d’une application multi-conteneur. Dans notre cas @oncletom nous propose de faire fonctionner hyphe sur 4 conteneurs Docker :
Pour cela vous devez ajouter une brique à Docker : docker-compose. Cette brique s’installe facilement en suivant les indications de la doc.
Une fois que c’est fait, vous pouvez vous rendre dans le répertoire dans lequel vous avez cloné hyphe et lancer l’installation des conteneurs grâce au Dockerfile.
cd github/hyphe/ docker-compose up
Il est temps d’aller prendre un café parce que l’installation prend un certain temps (15 min chez moi). La prochaine fois que vous lancerez l’application avec docker-compose up l’instanciation des composants se fera beaucoup plus rapidement.
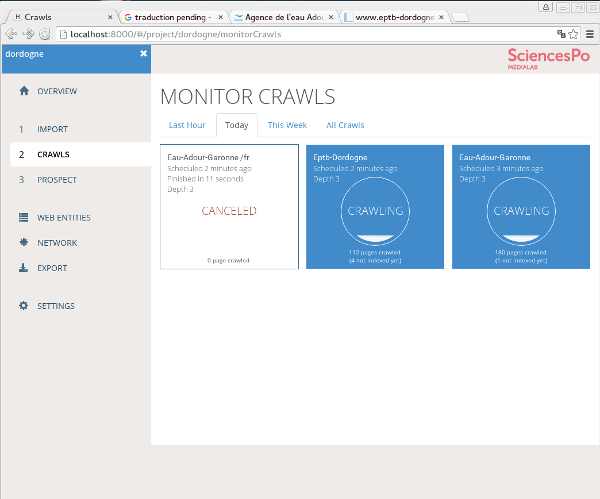
Vous accédez à l’application via votre navigateur (pour le moment, Firefox n’est pas supporter et vous devrez utiliser chrome ou chromium) à l’url suivante http://localhost:8000/#/login.
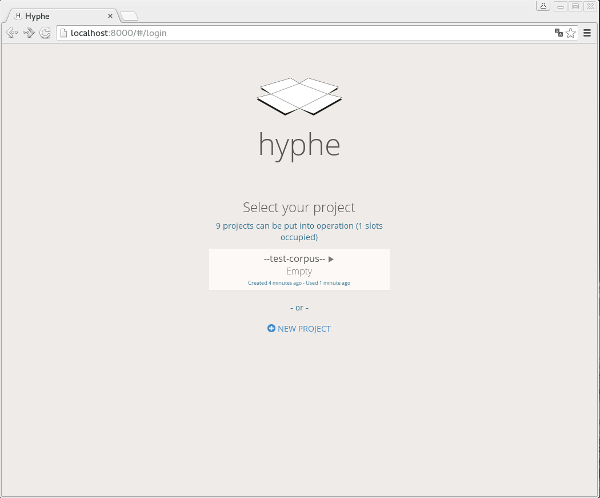 Vous pouvez explorer des projets « test », mais aussi et surtout vous lancer dans un nouveau projet et lâcher vos petits robot dans les méandres de la toile !
Vous pouvez explorer des projets « test », mais aussi et surtout vous lancer dans un nouveau projet et lâcher vos petits robot dans les méandres de la toile !
Note
Si vous avez besoin de relancer l’installation des conteneurs (s’il y a eu des maj de hyphe par exemple) vous pouvez reconstruire les conteneurs avec
docker-compose build
Fedora : un script post-install pour géographe
Depuis 2011, je prenais soin d’un petit archlinux. Je m’en occupais et faisais bien attention  à chaque mise à jour, ce qui se révélait parfois sportif, quand tout à coup, sans savoir pourquoi X11 ne répondait plus …
à chaque mise à jour, ce qui se révélait parfois sportif, quand tout à coup, sans savoir pourquoi X11 ne répondait plus …
Quand il a fallu rentrer en rédaction de thèse, la première mise à jour difficile m’a fait chercher un système stable… et j’ai essayé Fedora. Voilà maintenant 1 an que je suis donc dans le monde bleu, l’antichambre de Redhat… Et contre toute attente, j’en suis très satisfait. Mais voilà, peut-être à cause du manque de temps, je n’avais pas fait la migration de Fedora 20 à 21… donc il y a quelques semaines, les dernières mises à jour sont arrivées… et rien n’est plus triste que d’avoir un système figé…
Je me suis lancé dans la sauvegarde et la réinstallation du système, mais il faut bien avouer que le pire de tout est la phase de  post-install… les quelques jours où, à chaque besoin logiciel, il faut procéder à une installation.
post-install… les quelques jours où, à chaque besoin logiciel, il faut procéder à une installation.
En soi ce n’est pas forcément problématique, mais en cas de déplacement ou de perte de connexion … rien de plus frustrant. Aussi suis-je partie en quête d’un script de post-install Fedora. J’en ai trouvé un, proposé par Sam Hewitt sur gitHub.
Il ne me restait plus qu’à y ajouter quelques petites choses pour obtenir un beau script de post-install Fedora pour un géographe ! J’ai donc forké !
On y retrouve toutes les librairies spatiales qui vont bien (gdal, geos, proj4, etc), mais aussi R (et ses packages super comme rgdal, ggplot2, etc), et enfin une fresh build de texlive !
De quoi gagner du temps aux prochaines installations !